언젠가 기회가 되면 이 토픽에 대해서 발표를 하게 될지 모르겠다. 하지만 그때가 되기전에 한번 정리하고자 블로그에 올려본다.
재현성 있는 분석 혹은 리서치는 그 토픽에 대한 지속적인 연구를 가능하게 만드는 장점이 있다. 나도 역시 몇몇의 논문을 썼지만 코드나 데이터는 남아 있지 않다. 논문만 남았는데, 누군가 나의 논문 주제를 가지고 추가 연구를 하기가 정말 힘들어지는 단점이 남아있다. 금번 UseR! 2012 행사때도 이 주제가 큰 주제중에 하나였다. 바이오 정보학을 하시는 어떤 분께서 위에서 내가 이야기한 것과 같이 자신이 과거 누군가의 논문을 분석하는 과정이 얼마나 힘든 일이었는지 열변을 토하시며 발표하시는 것도 정말 인상 깊었다.
사실 논문 쓰기도 바쁘고 실험하기도 바쁜데, 이런것까지 신경써야 되나 하는 그런 이야기들을 많이 하는데, 개인적인 생각으로 이 재현성 부분을 정말 누구라도 재현이 실제 가능한 수준까지 논문에서 케어하고 있느냐가 논문이나 연구의 가치를 판단하는 큰 기준 중에 하나가 되어야 된다고 생각한다.
논문 이야기만 했지만 사실 분석업무도 누구든지 재현가능해야 된다고 생각한다. 분석팀이 있다면 팀원중에 누구라도 손쉽게 코드를 받고 데이터를 받아서 문서를 자동생성하고, 코드를 실행해 볼 수 있어야 된다. 흡사 이건 Git이나 SVN에서 코드를 받아서 make를 하는것과 굉장히 유사하다.
미국에서는 R이 학술/연구 영역에서 아주 활발하게 사용되고 있는데, R에서 이런 부분들을 케어하고 있는 여러 기능들이 최근에 많이 나오고 있는 상황이다. 이를 Literate programming이나 Reproducible research라는 용어로 표현한다.
데이터 분석 업무는 특정 플로우를 가지고 있으며 분석 과정은 이야기의 스토리를 만들어 가는것과 같다. 분석 과정 과정에는 분석가의 분석 견해가 들어가게 되고, 해석이 들어가고, 의미가 들어가야 한다. 따라서 분석은 R코드로만 설명될 수 있는 것들이 아니라 그 결과의 의미를 부여하는 과정이 필수인데, 이 때문에 분석이라는 업무 자체가 Literate programming과 맞아 떨어지게 되는 것이다.(문학적 프로그래밍, 번역 참 잘한거 같다. )
최근 팀 내부의 데이터 분석 문서화 및 코드관리에 대해서 어떻게 다룰까 많은 논의가 있었는데, 결국 재현성의 관점에서 많은 이야기가 나오게 되었고, 전통적인 Git, SVN을 데이터 분석에서도 사용하고 코드와 문서를 단 하나의 파일로 관리하고 그 파일에서 자동으로 문서생성과 코드실행이 되고 문서도 여러 포맷으로 변환 가능하게끔 분석 프로세스를 정하는 것으로 방향을 잡았다.
그리고 이 중심에 있는게 RStudio, R Markdown, Git, Pandoc 그리고 knitr이다.
이것들이 활용이 되는 과정은 아래와 같다.
먼저 단일 Rmd파일로 R코드와 Markdown 문학적 프로그래밍을 한다. 물론 이 편집 기능은 RStudio에서 제공하고 있다. Rmd 파일을 순수 Markdown 문서로의 변환은 knitr이 담당한다. knitr은 Rmd파일에 있는 R 코드를 실행하고 플로팅 이미지를 생성해 Markdown문서에 링크한다. 그리고 Pandoc으로 생성된 Markdown 문서를 다양한 포맷으로 변환한다. 실제 팀 내부에서는 Markdown문서를 MS Word파일로 변환하는 것을 보고 상당한 감명을 받았다. 왜냐면 생성된 문서에서 코드 하이라이팅과 링크 그리고 이미지가 그대로 하나의 정리된 문서로 생성되었기 때문이다. 물론 Pandoc을 사용하면 Markdown문서를 Slide로 변환한이 가능하다. 만일 분석 문서를 발표자료로 만든다면 10초도 안걸리고 Markdown으로 관리되는 문서를 기반으로 슬라이드 생성이 가능해진다.
그리고 결국 우리는 Rmd파일 하나만 관리하면 되는데 이 문서가 코드가 함께 포함된 문서여서 Git을 기반으로 이력관리를 하면 된다. 재밋게도 GitHub에서는 Markdown문서 뷰어가 내장되어 있어 md 확장자를 가진 파일을 자동으로 변환해 보여준다. 이렇게 되면 GitHub을 분석 문서 공유처로 활용해도 된다.
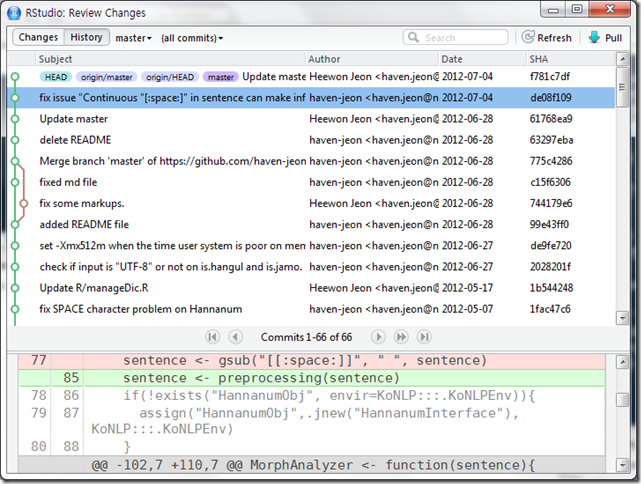
그리고 우리의 사랑스러운 RStudio에서 SVN, Git을 지원하고 있다.
아래는 KoNLP 패키지의 Git History이다.
물론 여기서 빠진게 데이터이다. 데이터는 GitHub에서 관리하는 건 비추다. 왜냐면 대부분 대용량의 데이터를 사용해 분석하기 때문하기 때문이고 데이터의 경우 보안 이슈가 빠지지 않고 나오는데 이를 Git으로 케어하는건 맞지 않기 때문이다. 따라서 데이터는 별도의 사내의 파일서버를 활용하거나 웹서버 등을 활용하는 것을 추천하고 R코드에서는 원격으로 읽어들일 수 있게 하면 된다.
참, 그리고 R Markdown 문서에는 sessionInfo()와 set.seed() 코드가 반드시 들어가야 된다.
나의 현재 세션정보는 아래와 같다. 이 결과 텍스트를 문서를 보는 사람이 확인하고 데이터와 코드가 어떤 환경에서 돌았는지 알 수 있게 된다. 그리고 랜덤함수종류를 사용했다면 set.seed(XXX)와 같은 코드로 랜덤 넘버의 재현성을 높여주는게 좋다.
> sessionInfo() R version 2.15.1 (2012-06-22) Platform: x86_64-pc-mingw32/x64 (64-bit) locale: [1] LC_COLLATE=Korean_Korea.949 LC_CTYPE=Korean_Korea.949 [3] LC_MONETARY=Korean_Korea.949 LC_NUMERIC=C [5] LC_TIME=Korean_Korea.949 attached base packages: [1] stats graphics grDevices utils datasets methods [7] base other attached packages: [1] ggplot2_0.9.1 KoNLP_0.73 bitops_1.0-4.1 rJava_0.9-3 loaded via a namespace (and not attached): [1] colorspace_1.1-1 dichromat_1.2-4 digest_0.5.2 [4] grid_2.15.1 labeling_0.1 MASS_7.3-19 [7] memoise_0.1 munsell_0.3 plyr_1.7.1 [10] proto_0.3-9.2 RColorBrewer_1.0-5 reshape2_1.2.1 [13] scales_0.2.1 stringr_0.6 tools_2.15.1
빅 데이터 분석의 경우 상당한 전처리 코드와 분석 코드를 만들게 되는데, 이 코드가 반드시 생각한대로 동작한다는 보장이 없다. 이렇게 말하는 이유가 뭐냐면, 사람이 만든 코드는 항상 버그가 숨어 있기 때문이다. 그렇다고 분석가들도 코드리뷰를 해야 된다고 강제하고 싶지는 않지만 코드리뷰 과정이 있다면 분석 결과에 대해서 상당히 신뢰할 수 있게 된다. 그리고 만일 분석이 재현성 있게 관리가 된다면 업무 담당자 뿐만 아니라 다른 분석가들도 쉽게 분석 리소스에 접근해 확인이 가능하여 투명한 분석 리소스 관리가 가능해지고, 접근이 쉬워 여러 사람의 눈으로 확인이 쉽게 된다면 버그를 발견할 확률도 높아지게 된다. 이런 버그나 오류를 발견하는 횟수가 늘어나면 분석 결과의 신뢰성도 높아진다. 물론 이렇게 관리한다면 분석가들이 맘 편하게 코드 commit을 하지 못하게 되겠지만 말이다. ^^;
현재로서 분석/리서치를 한다면 R을 사용하는게 가장 탁월한 선택이라고 자신있게 말하고 싶다. 왜냐면 이미 내가 위에서 말한 재현성, 분석 관리 이슈를 가장 잘 케어하고 있기 때문이다.
ps. 언제 이 주제로 공개 발표를 할 수 있으면 좋겠는데, 다들 빅 데이터 발표만 해달라고 하시니 언제가 될지는 기약하기 힘들거 같다. ㅡㅡ;